刘毓文(联合培养导师)
2023-03-08 17:51:34 浏览数:0

个人简介: 刘毓文,研究员、博士生导师。入选国家重大人才工程(青年)、广东省高层次人才(青年)、中国农业科学院“农科英才”领军人才B类、深圳市高层次人才B类,深圳市“青年五四奖章”获得者。2006年于清华大学本科毕业,2014年在芝加哥大学取得博士学位。刘毓文研究员在基因组和表观遗传学领域有着十多年的科学研究经历,在学术研究上擅长开发并利用实验和计算方法,从非编码顺式调控区入手解析复杂性状的遗传基础,以及基于细胞调控线路设计的合成生物学育种。
以通讯作者(含共同通讯)作者在Genome Research,Nature Communications, Nucleic Acids Research, Advanced Science等高水平杂志上发表多篇文章, Google Scholar总引用1800多次。开发了WHG-STARR-Seq和Ss-STARR-Seq实验方法,分别首次实现了哺乳动物全基因组增强子和沉默子的高通量直接定量;开发了等位基因特异性allele-specific STARR-Seq,首次测定了上万GWAS关联性SNP对增强子功能影响;开发了统计遗传学算法TADA-Annotation,首次将整合增强子功能性变异等多种功能基因组注释用于挖掘挖掘复杂性状因果基因的方法拓展到新生突变。开发了DREAM算法实现基于AI的顺式调控元件从头设计,在多个物种中获得了超过目前最强活性的增强子序列,产生了首个基因组中整合入AI设计增强子的成体动物。在动物遗传育种领域,率先利用等位基因特异性STARR-Seq,从增强子功能性变异为切入点,挖掘了生猪产肉相关性状的潜在因果调控型变异和功能基因。开发了基于靶向二代测序的伴侣动物基因检测液相芯片,可以通过品系鉴定,遗传病筛查和亲缘关系鉴定提升遗传改良效率,获批三项国家专利,获得产业投资400万元进行产业转化。开发了基于超低覆盖度全基因组测序数据的基因型分型计算方法,领导团队开发了适用于二倍体农业动物和作物的高效低成本基因型分型和基因组选择平台,首次实现了单个动物囊胚的全基因组SNP准确分析,为胚胎基因组选择提供了重要工具,获得首届广东省种业科技大比武总决赛十强,2021年深圳市创新创业大赛大鹏赛区农业赛道第一名,并以此平台依托基因组所孵化了中农芯跃分子育种公司,该高效低成本基因组选择平台于2023年获得《中国科学报》、《中央人民广播电台大湾区之声》等多家媒体的专题报道。主持十四五国家重点研发计划课题、国家自然科学青年科学基金项目、国家自然科学面上项目等国家级项目4项,主持深圳市科创委项目1项,核心参与了广东省基础与应用基础研究基金项目、中国农业科学院科技创新工程重大科研计划、十四五重点研发计划等多项项目。
研究方向:动物遗传育种与繁殖
学习工作经历:
[1]教育经历:
2006-2014 芝加哥大学获得生物学博士学位(研究方向基因组学) 导师:Kevin White教授
2002-2006 清华大学生物系获学士学位 导师:裴端卿教授
[2]工作经历:
2021-至今 动物基因组研究中心副主任(中国农业科学院农业基因组研究所)
2019-至今 研究员(中国农业科学院农业基因组研究所)
2015-2018 博士后(芝加哥大学人类遗传系)导师贺信教授
2016-2018 芝加哥大学生物信息学和计算生物学暑期课程讲师
2014-2015 博士后(基因组和系统生物学研究中心) 导师:Kevin White教授
科学研究:
主要专注于开发新的实验和计算方法,研究转录调控中的增强子定位和功能,非编码区DNA变异如何通过影响增强子功能而影响生物性状,以及如何通过人工设计调控增强子等调控元件改变生物性状,方法具有普遍适用性,同时在解析猪产肉性状遗传机制解析中进行深入应用。具体研究方向包括:
1.结合基因型数据,表型数据和多组学功能注释数据,解析GWAS信号和选择进化信号,从而挖掘影响性状的重要调控元件和基因;
2.开发高通量实验和计算方法,在动物体内和单细胞层面,鉴定影响增强子功能的DNA变异,并以此为切入点,挖掘影响性状的关键基因;
3.开发实验和计算方法,结合高通量调控元件数据进行调控元件序列底层逻辑的解析,优化和设计新一代调控元件和细胞线路,通过合成生物学手段为性状改良提供基因功能模块;
4.开发低成本高密度的新一代基因型检测方法,开发结合多组学数据和深度学习的全基因组选育方法和精细定位方法,协助推进畜牧动物品种选育和性状改良。
教学授课情况:
1.实用生物信息技术 专业课 理论课 24学时
2.分子遗传学 专业课 理论课 8学时
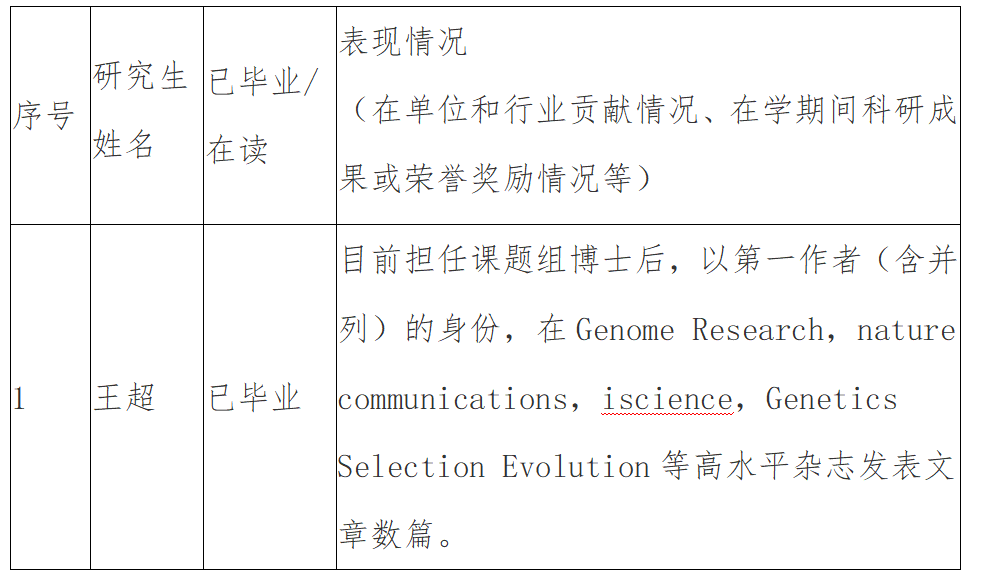
获奖及社会兼职等情况:
获奖情况:
1. 2021 深圳市青年五四奖章
2. 2021 首届广东省农业科技创新大比武总决赛十强
3. 2022 深圳市创新创业大赛大鹏赛区一等奖海洋农业食品综合赛道第一名
4.2022 第二十四届中国国际高新技术成果交易会优秀产品奖
社会兼职:
中国生物信息学学会青年工作委员会负责人
农业农村部畜禽生物组学重点实验室学术委员会委员
国家特种经济动物科技创新联盟第二届理事会副理事长
联系方式:邮箱liuyuwen@caas.cn
- 上一篇: 陶聪(联合培养导师)
- 下一篇: 孔思远(联合培养导师)


